arpack:直接使用 scipy 库的 sparse SVD 实现,和 randomized 的适用场景类似
import numpy as np import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import Axes3D from sklearn.decomposition import PCA from sklearn.datasets.samples_generator import make_blobs X, y = make_blobs(n_samples=10000, n_features=3, centers=[[3, 3, 3], [0, 0, 0], [1, 1, 1], [2, 2, 2]], cluster_std=[0.2, 0.1, 0.2, 0.2], random_state=9) fig = plt.figure() plt.scatter(X_new[:, 0], X_new[:, 1], marker='o') plt.show()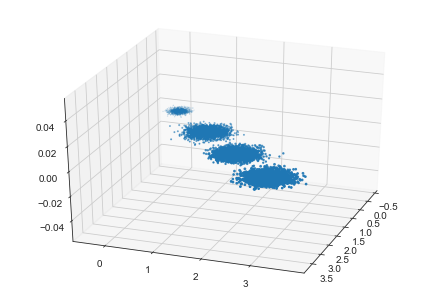
这样我们就实现了三维到二维的转换,可以把结果可视化:
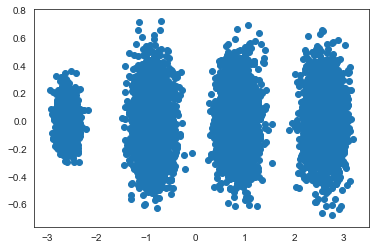
PCA算法优点
1,仅仅需要以方差衡量信息量,不受数据集以外的因素影响
2,各主成分之间正交,可消除原始数据成分间的相互影响的因素
3,计算方法简单,主要运算时特征值分解,易于实现
4,它是无监督学习,完全无参数限制的。
PCA算法缺点
1,主成分各个特征维度的含义具有一定的模糊性,不如原始样本特征的解释性强
2,方差小的非主成分也可能含有对样本差异的重要信息,因降维丢弃可能对后续数据处理有影响。
参考https://github.com/fengdu78/lihang-code
https://finthon.com/python-pca/
https://www.cnblogs.com/pinard/p/6239403.html

